El SARS-CoV‑2 es el séptimo miembro de los Coronaviridae que se sabe que infectan a los humanos. Tres de estos virus, SARS CoV‑1, MERS y SARS-CoV‑2, pueden causar enfermedades graves; cuatro, HKU1, NL63, OC43 y 229E, están asociados con síntomas respiratorios leves. Aquí revisamos lo que se puede deducir sobre el origen y la evolución temprana del SARS-CoV‑2 a partir del análisis comparativo de los datos disponibles sobre la secuencia del genoma. En particular, ofrecemos una perspectiva de las notables características del genoma del SARS-CoV‑2 y discutimos los escenarios en los que estas características podrían haber surgido. Es importante señalar que este análisis proporciona pruebas de que el SARS-CoV‑2 no es una construcción de laboratorio ni un virus manipulado a propósito.
La comparación genómica de los virus alfa y betacoronavirus (familia Coronaviridae ) que se describe a continuación identifica dos características notables del genoma del SARS-CoV‑2: i) Sobre la base de la modelización estructural y los primeros experimentos bioquímicos, el SARS-CoV‑2 parece estar optimizado para unirse al receptor humano de la ECA2; ii) la proteína de punta altamente variable (S) del SARS-CoV‑2 tiene un sitio de clivaje polibásico (furina) en el límite S1 y S2 mediante la inserción de doce nucleótidos. Además, este evento condujo a la adquisición de tres glicanos ligados al O pronosticado alrededor del sitio de clivaje polibásico.
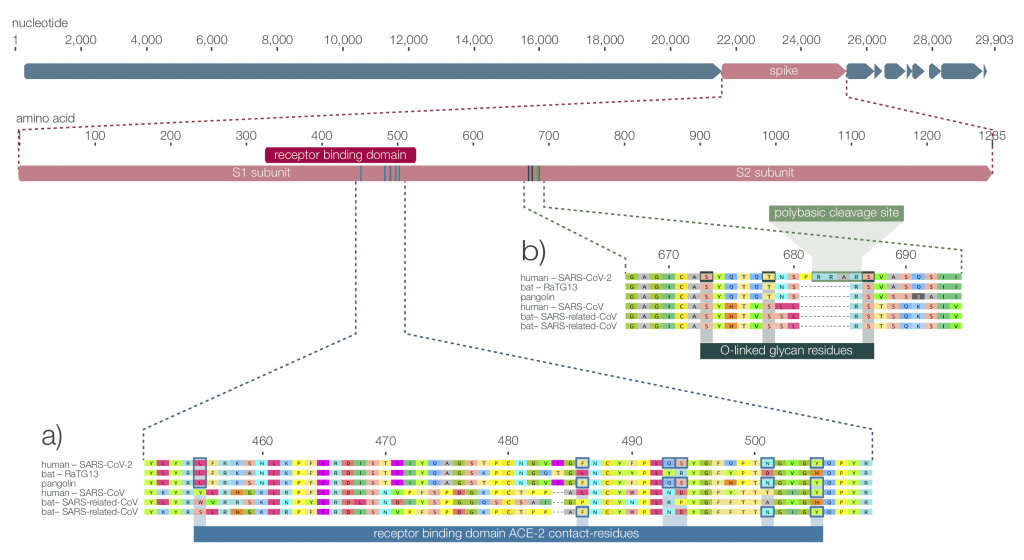
Mutaciones en el dominio de unión de receptores del SARS-CoV‑2
El dominio de unión al receptor (RBD) en la proteína de punta del SARS-CoV y los coronavirus relacionados con el SARS es la parte más variable del genoma del virus. Seis residuos en el RBD parecen ser críticos para la unión al receptor humano ACE2 y para determinar el rango del huésped1. Usando coordenadas basadas en la cepa de Urbani del SARS-CoV, son Y442, L472, N479, D480, T487, y Y4911. Los residuos correspondientes en el SARS-CoV‑2 son L455, F486, Q493, S494, N501, y Y505. Cinco de estos seis residuos están mutados en el SARS-CoV‑2 en comparación con su virus más estrechamente relacionado, el RaTG13 muestreado de un murciélago de Rhinolophus affinis, al que es ~96% idéntico2 (Figura 1a). Sobre la base de la modelización1 y de experimentos bioquímicos3,4 , el SARS-CoV‑2 parece tener un RBD que puede unirse con alta afinidad al ACE2 de humanos, primates no humanos, hurones, cerdos y gatos, así como de otras especies con alta homología de receptores1. Por el contrario, el SARS-CoV‑2 puede unirse de manera menos eficiente a la IECA2 en otras especies asociadas con virus similares al SARS, incluidos roedores y civetas1.
La fenilalanina (F) en el residuo 486 de la proteína S del SARS-CoV‑2 corresponde a L472 en la cepa Urbani del SARS-CoV. Cabe destacar que en los experimentos de cultivo celular del SARS-CoV el L472 muta a fenilalanina (L472F), que se prevé que sea óptima para la unión de la RBD del SARS-CoV al receptor humano ACE. Sin embargo, una fenilalanina en esta posición también está presente en varios CoV similares al SARS de murciélagos (Figura 1a). Si bien estos análisis sugieren que el SARS-CoV‑2 puede ser capaz de unir el receptor humano de la ECA2 con gran afinidad, no se prevé que la interacción sea óptima. Además, varios de los residuos clave en el RBD del SARS-CoV‑2 son diferentes de los descritos anteriormente como óptimos para la unión del receptor humano de IECA6. En contraste con estas predicciones computacionales, estudios recientes sobre la unión indican que el SARS-CoV‑2 se une con una alta afinidad al ACE humano. Así pues, el pico del SARS-CoV‑2 parece ser el resultado de la selección de un IECA humano o similar al humano que permite que surja otra solución óptima de unión. Esta es una fuerte evidencia de que el SARS-CoV‑2 no es el producto de la ingeniería genética.
Sitio de división polibásica y glicanos ligados al O
La segunda característica notable del SARS-CoV‑2 es un sitio de clivaje polibásico predictivo (RRAR) en la proteína de punta en la unión de S1 y S2, las dos subunidades de la proteína de punta (figura 1b) . Además de dos argininas básicas y una alanina en el sitio de clivaje, también se inserta una prolina principal; por lo tanto, la secuencia totalmente insertada es la ARP (Figura 1b). Se prevé que el fuerte giro creado por la inserción de la prolina resulte en la adición de glicanos ligados al O a los S673, T678 y S686 que flanquean el sitio de clivaje polibásico. No se ha observado anteriormente un sitio de división polibásica en los betacoronavirus del linaje B relacionados y es una característica única del SARS-CoV‑2. Algunos betacoronavirus humanos, entre ellos el HCoV-HKU1 (linaje A), tienen sitios de división polibásica, así como glicanos vinculados al O pronosticados cerca del sitio de división S1/S2.
Si bien se desconoce la consecuencia funcional del sitio de clivaje polibásico en el SARS-CoV‑2, los experimentos con el SARS-CoV han demostrado que la ingeniería de ese sitio en la unión S1/S2 mejora la fusión célula-célula pero no afecta a la entrada del virus10. Los sitios de clivaje polibásico permiten un clivaje efectivo por la furina y otras proteasas, y pueden adquirirse en la unión de las dos subunidades de la proteína hemaglutinina (HA) de los virus de la gripe aviar en condiciones que seleccionan para una rápida replicación y transmisión del virus (por ejemplo, poblaciones de pollos muy densas). La HA cumple una función similar a la de la proteína S del coronavirus en la fusión célula-célula y la entrada del virus. La adquisición de un sitio de división polibásica en la HA, ya sea por inserción o recombinación, convierte los virus de la gripe aviar de baja patogenicidad en formas altamente patógenas. También se ha observado la adquisición de sitios de división polibásica por el virus de la gripe HA después del paso forzado repetido en cultivos celulares o a través de animales. Análogamente, un aislado avirulento del virus de la enfermedad de Newcastle se convirtió en altamente patógeno durante el paso en serie en pollos mediante la adquisición incremental de un sitio de división polibásica en la unión de sus subunidades de proteína de fusión. La función potencial de los tres glicanos ligados al O pronosticado es menos clara, pero podrían crear un “dominio similar a la mucina” que protegería los posibles epítopos o residuos clave en la proteína de punta del SARS-CoV‑2. Se requieren análisis bioquímicos o estudios estructurales para determinar si se utilizan o no los sitios de glicanos ligados al O previstos.
Teorías sobre los orígenes del SARS-CoV‑2
Es improbable que el SARS-CoV‑2 haya surgido mediante la manipulación en laboratorio de un coronavirus existente relacionado con el SARS. Como se ha señalado anteriormente, el RBD del SARS-CoV‑2 está optimizado para la unión del receptor humano ACE2 con una solución de unión eficiente diferente a la que se habría predicho. Además, si se hubiera realizado una manipulación genética, cabría esperar que se hubiera utilizado uno de los diversos sistemas genéticos inversos disponibles para los betacoronavirus. Sin embargo, no es así, ya que los datos genéticos muestran que el SARS-CoV‑2 no se deriva de ninguna espina dorsal de virus previamente utilizada.
En su lugar, proponemos dos hipótesis que pueden explicar de manera plausible el origen del SRAS-CoV‑2:
- la selección natural en un huésped animal no humano antes de la transferencia zoonótica, y
- la selección natural en los seres humanos después de la transferencia zoonótica. También se examina si la selección durante el paso por el cultivo podría haber dado lugar a las mismas características observadas.
Selección en un animal huésped
Dado que muchos de los primeros casos de COVID-19 estaban relacionados con el mercado de mariscos y vida silvestre de Huanan en Wuhan, es posible que una fuente animal estuviera presente en este lugar. Dada la similitud del SARS-CoV‑2 con los CoV similares al SARS de los murciélagos, en particular el RaTG13, es plausible que los murciélagos sirvan como reservorios de hospedaje del SARS-CoV‑2. Sin embargo, es importante señalar que anteriores brotes de betacoronavirus en seres humanos implicaron la exposición directa a animales distintos de los murciélagos, incluidas las civetas (SARS) y los camellos (MERS), que son portadores de virus que son genéticamente muy similares al SARS-CoV‑1 o al MERS-CoV, respectivamente. Por analogía, los virus estrechamente relacionados con el SARS-Cov‑2 pueden estar circulando en una o más especies animales. Los análisis iniciales indican que los pangolines malayos ( Manis javanica ) importados ilegalmente a la provincia de Guangdong contienen un CoV similar al SARS-CoV-218. Aunque el virus de los murciélagos RaTG13 sigue siendo el pariente más cercano al SARS-CoV‑2 en todo el genoma, el CoV del pangolín malayo es idéntico al SARS-CoV‑2 en los seis residuos clave del RBD (figura 1). Sin embargo, aún no se ha identificado ningún pangolín CoV que sea lo suficientemente similar al SARS-CoV‑2 en todo su genoma como para soportar una infección humana directa. Además, el pangolín CoV no lleva una inserción en el sitio de clivaje polibásico. Para que un virus precursor adquiera el sitio de clivaje polibásico y las mutaciones en la proteína de punta adecuada para la unión del receptor ACE2 humano, un animal huésped probablemente tendría que tener una alta densidad de población — para permitir que la selección natural proceda de manera eficiente — y un gen ACE2 que sea similar al ortólogo humano. La caracterización ulterior de los CoV en pangolines y otros animales que puedan albergar virus similares al SARS-CoV debería ser una prioridad de salud pública.
Adaptación críptica a los humanos
También es posible que un progenitor del SARS-CoV‑2 haya saltado de un animal no humano a los humanos, con las características genómicas descritas anteriormente adquiridas mediante la adaptación durante la posterior transmisión de humano a humano. Suponemos que una vez adquiridas estas adaptaciones (ya sea juntas o en serie) permitiría el despegue del brote, produciendo un grupo suficientemente grande e inusual de casos de neumonía para activar el sistema de vigilancia que finalmente lo detectó.
Todos los genomas del SARS-CoV‑2 secuenciados hasta ahora tienen el RBD bien adaptado y el sitio de clivaje polibásico, y por lo tanto se derivan de un antepasado común que tenía estas características. La presencia de un RBD en las pangolinas que es muy similar al del SARS-CoV‑2 significa que probablemente ya estaba presente en el virus que saltó a los humanos, aunque todavía no tenemos el virus progenitor no humano exacto. Esto deja que la inserción del sitio de división polibásica ocurra durante la transmisión de humano a humano. Siguiendo el ejemplo del gen HA del virus de la gripe A, se requiere un evento específico de inserción o recombinación para permitir el surgimiento del SARS-CoV‑2 como un patógeno epidémico.
Las estimaciones del momento en que se produce el antepasado común más reciente (tMRCA) del SARS-CoV‑2 utilizando los datos de secuencias genómicas actualmente disponibles apuntan a la aparición del virus entre finales de noviembre y principios de diciembre de 2019, lo que es compatible con los primeros casos confirmados retrospectivamente. Por lo tanto, este escenario supone un período de transmisión no reconocida en los seres humanos entre el evento inicial de transferencia zoonótica y la adquisición del sitio de división polibásica. Podría darse una oportunidad suficiente si hubiera habido muchos eventos zoonóticos anteriores que hubieran producido cadenas cortas de transmisión entre humanos (las llamadas “cadenas de tartamudez”) durante un período prolongado. Esta es esencialmente la situación para el MERS-CoV en la Península Arábiga donde todos los casos humanos son el resultado de repetidos saltos del virus desde los camellos dromedarios, produciendo infecciones únicas o cadenas cortas de transmisión que eventualmente se resuelven. Hasta la fecha, después de 2.499 casos en 8 años, no ha surgido ninguna adaptación humana que haya permitido al MERS-CoV arraigarse en la población humana.
¿Cómo podríamos probar si la propagación críptica del SARS-CoV‑2 permitió la adaptación humana? Los estudios metagenómicos de las muestras de suero almacenadas podrían proporcionar información importante, pero dado el período relativamente breve de viremia, tal vez sea imposible detectar la circulación del SARS-CoV‑2 de bajo nivel en las muestras históricas. Los estudios serológicos retrospectivos podrían ser informativos y ya se han realizado algunos de esos estudios. En uno de ellos se determinó que los comerciantes de importación de animales tenían una seropositividad del 13% a los coronavirus, mientras que en otro se observó que el 3% de los residentes de una aldea del sur de China eran seropositivos a esos virus. Curiosamente, 200 residentes de Wuhan no mostraron serorreactividad a los coronavirus. Sin embargo, lo más importante es que esos estudios no pudieron distinguir si las respuestas serológicas positivas se debían a una infección previa con el virus del SARS-CoV‑1 o ‑2. Deberían realizarse más estudios serológicos retrospectivos para determinar el alcance de la exposición humana anterior a los betacoronavirus en diferentes zonas geográficas, en particular utilizando ensayos que permitan distinguir entre múltiples betacoronavirus.
Selección durante el paso
Las investigaciones básicas sobre el paso de coronavirus similares al SARS de los murciélagos en cultivos celulares y/o modelos animales han estado en curso en el BSL‑2 durante muchos años en múltiples laboratorios de todo el mundo. También hay casos documentados de adquisición del SARS-CoV‑1 por personal de laboratorio que trabaja en la contención del BSL‑2. Por consiguiente, debemos considerar la posibilidad de una liberación deliberada o inadvertida de SARS-CoV‑2. En teoría, es posible que el SARS-CoV‑2 haya adquirido el sitio de las mutaciones del RBD observadas durante la adaptación al paso en el cultivo celular, como se ha observado en estudios con el SARS-CoV5 así como con el MERS-CoV31. Sin embargo, la adquisición del sitio de división polibásica o de los glicanos ligados al O — si son funcionales — se opone a este escenario. Los nuevos sitios de división polibásica sólo se han observado después del paso prolongado del virus de la gripe aviar de baja patogenicidad en cultivos celulares o animales. Además, la generación del SARS-CoV‑2 por cultivo celular o paso de animales habría requerido el aislamiento previo de un virus progenitor con una similitud genética muy alta. La generación posterior de un sitio de división polibásica habría requerido entonces un intenso programa de paso en cultivos celulares o animales con un receptor ACE‑2 similar al de los seres humanos (por ejemplo, hurones). También es cuestionable si la generación de los glicanos ligados al O se habría producido en el paso por el cultivo celular, ya que esas mutaciones suelen sugerir la participación de un sistema inmunológico que no está presente in vitro.
Conclusiones
En medio de la emergencia de salud pública mundial de COVID-19 es razonable preguntarse por qué importan los orígenes de la epidemia. Una comprensión detallada de cómo un virus animal saltó las fronteras de las especies para infectar a los humanos de manera tan productiva ayudará en la prevención de futuros eventos zoonóticos. Por ejemplo, si el SARS-CoV‑2 se preadaptó en otra especie animal, entonces estamos en riesgo de futuros eventos de re-emergencia, incluso si la actual epidemia está controlada. En cambio, si el proceso de adaptación que describimos se produjo en seres humanos, entonces, aunque tengamos repetidas transferencias zoonóticas, es improbable que despeguen a menos que se produzca la misma serie de mutaciones. Además, la identificación de los parientes animales más cercanos del SARS-CoV‑2 será de gran ayuda para los estudios de la función del virus. De hecho, la disponibilidad de la secuencia de murciélagos RaTG13 facilitó el análisis genómico comparativo realizado aquí, ayudando a revelar las mutaciones clave en el RBD así como la inserción del sitio de clivaje polibásico.
Las características genómicas aquí descritas pueden explicar en parte la infecciosidad y la transmisibilidad del SARS-CoV‑2 en los seres humanos. Aunque las pruebas genómicas no apoyan la idea de que el SARS-CoV‑2 es una construcción de laboratorio, actualmente es imposible probar o refutar las otras teorías de su origen descritas aquí, y no está claro si los datos futuros ayudarán a resolver este problema. La identificación de la fuente animal no humana inmediata y la obtención de secuencias de virus a partir de ella sería la forma más definitiva de revelar los orígenes del virus. Además, sería útil obtener más datos genéticos y funcionales sobre el virus, incluidos los estudios experimentales de la unión de los receptores y el papel del sitio de división polibásica y los glicanos vinculados con el O previsto. La identificación de un posible huésped intermedio del SARS-CoV‑2, así como la secuenciación de los casos muy tempranos, incluidos los que no están relacionados con el mercado de Wuhan, sería igualmente muy informativa. Independientemente de la forma en que se originó el SARS-CoV‑2, es evidente que la vigilancia continua de la neumonía en los seres humanos y otros animales es de suma importancia.
Kristian G. Andersen es miembro del Departamento de Inmunología y Microbiología, el Instituto de Investigación Scripps, La Jolla, EE.UU.
Comparte esta publicación
Suscríbete a nuestro boletín
Recibe toda la actualidad en cultura y ocio, de la ciudad de Valencia